SplitsTree
SplitsTree é um programa popular para inferir árvores filogenéticas ou, mais genericamente, redes filogenéticas de vários tipos de dados, tais como a alinhamento de sequências, uma matriz de distâncias ou um conjunto de árvores[1][2].
| SplitsTree | |
|---|---|
| Desenvolvedor | Daniel Huson e David Bryant |
| Versão estável | 4.10 (2008) |
| Sistema operacional | Mac OS X, Windows, Unix-like |
| Gênero(s) | Bioinformática |
| Licença | livre para uso mas não é de código aberto |
| Página oficial | SplitsTree |
SplitsTree implementa métodos publicados tais como a decomposição Split neighbor-net, redes de consenso, os métodos de super-redes ou métodos de hibridização por computação ou de redes de recombinação simples.
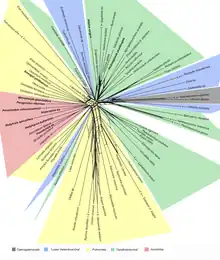
Um exemplo de uma rede filogenética neighbor-net gerada pelo SplitsTree v4.6.
Ver também
Ligações externas
- Página oficial do SplitsTree
- Lista de software de filogenia, hopedado na University of Washington
Referências
- Dress, A.; Huber, K. T.; Moulton, V. (2001). «Metric spaces in pure and applied mathematics» (PDF). Documenta Mathematica. p. 121–139
- Huson, D. H.; Bryant, D. (2006). «Application of Phylogenetic Networks in Evolutionary Studies». Mol. Biol. Evol. 23 (2). p. 254–267. PMID 16221896. doi:10.1093/molbev/msj030
This article is issued from Wikipedia. The text is licensed under Creative Commons - Attribution - Sharealike. Additional terms may apply for the media files.